×
Activités de Recherche
Durant les dernières décennies, la quantité d'informations récupérées et stockées a explosé atteignant aisément les giga/tera -octets ou plus encore, et l'exploitation et l'analyse d'une telle masse de données constituent un réel défi.
Ont ainsi émergé les problématiques d'analyse, de traitement et d'exploitation de données massives, généralement regroupées sous l'étiquette problèmatique Big Data.
Le traitement de ces données soulève énormément de problèmes, d'une part à cause de leurs volumes, mais aussi à cause de l'architecture de stockage/traitement.
En effet, une architecture Big Data est généralement constituée de plusieurs espaces de stockage et de plusieurs machines qui accèdent et traitent les données de façon distribuée.
Ainsi, le principe même de traitements et d'analyses Big Data implique qu'il y ait une contrainte d'éloignement géographique forte entre les données (stockage et traitement) et l'utilisateur (analyse), se posent donc les problèmes d'accès, de traitements, de transferts et de délais nécessaires pour permettre ces opérations.
Par ailleurs, la complexité des données constitue en elle-même un défi pour l'analyse et l’interprétation, qu'elles soient massives ou non.
La visualisation d'information est une des solutions pour répondre à cette problématique et vise à représenter sous forme visuelle, graphique et parfois interactive les données afin d'améliorer la capacité humaine à les explorer et en extraire une information.
Cependant, utiliser une telle masse d'information dans son intégralité pose le problème de l'utilisabilité de la représentation obtenue (chevauchement d'informations, encombrement) et de la difficulté de compréhension que cela implique pour un utilisateur.
Ainsi, obtenir une représentation juste, utilisable et efficace de données massives constitue un réel défi pour l'exploration interactive et l'analyse de l'information qu'elles véhiculent.
Dans mon activité de recherche, je m'intéresse aux techniques de visualisation abstraite et notamment à leur adaptation dans le cadre de données massives pour en faciliter l'exploration et l'interaction, en particulier pour des données issues du web analytics.
Pour répondre à ces problèmes, les solutions abordées s'appuient sur la mise en place d'abstractions et de métaphores visuelles adaptées aux données massives et permettant la mise en évidence de corrélations et la présence (ou l'absence) de motifs particuliers dans les données.
Afin d'aider les utilisateurs dans leurs explorations, il est indispensable de leur fournir des outils d'interaction adaptés à ces nouvelles techniques de visualisation tels que l'exploration de représentation multi-échelles, le détail à la demande, etc. qui soulèvent des problèmes similaires.
Pour cela, les différents outils et systèmes produits sont créés sur la base de deux principes: utiliser des langages permettant une exploitation distante des données (technologies web) tout en répondant aux impératifs de performances dans les traitements (langage de programmation graphique OpenGL principalement).
Les axes de recherche sont donc divisés en deux parties: la visualisation de données abstraites et l'application pour les données massives et deux types de données ont été envisagées à l'heure actuelle\,: les données relationnelles, qui représentent l'ensemble des relations entre des entités, et les données multi-dimensionnelles, qui représentent quant à elles les attributs d'un ensemble d'entités.
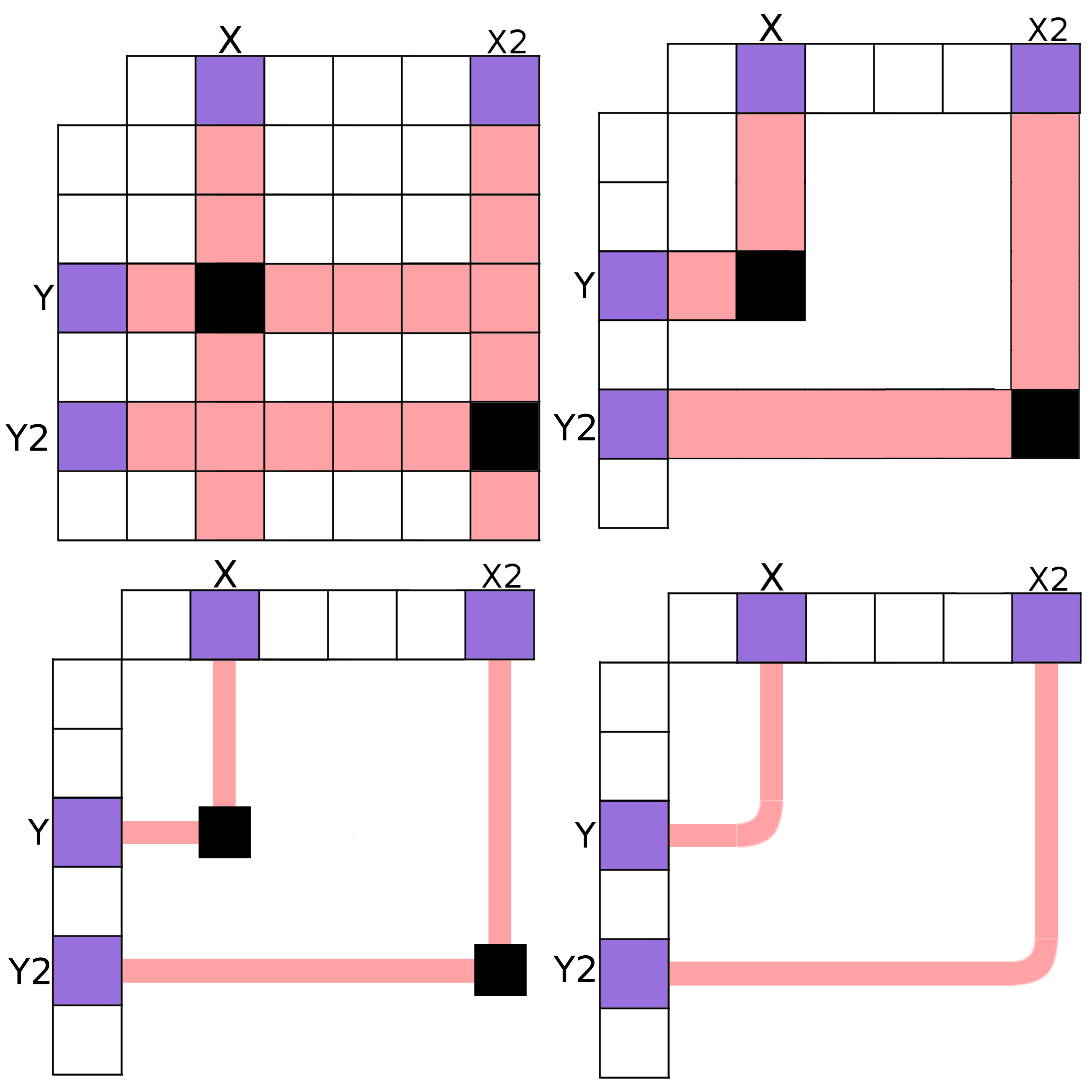
Un premier aspect de recherche a été consacré à l'étude de l'impact que l'encodage visuel des arêtes dans une des représentations classiques de graphe peut avoir sur les performances utilisateurs, à la fois sur un ensemble de tâches simples mais aussi sur une tâche complexe fréquente en analyse de graphe [1].
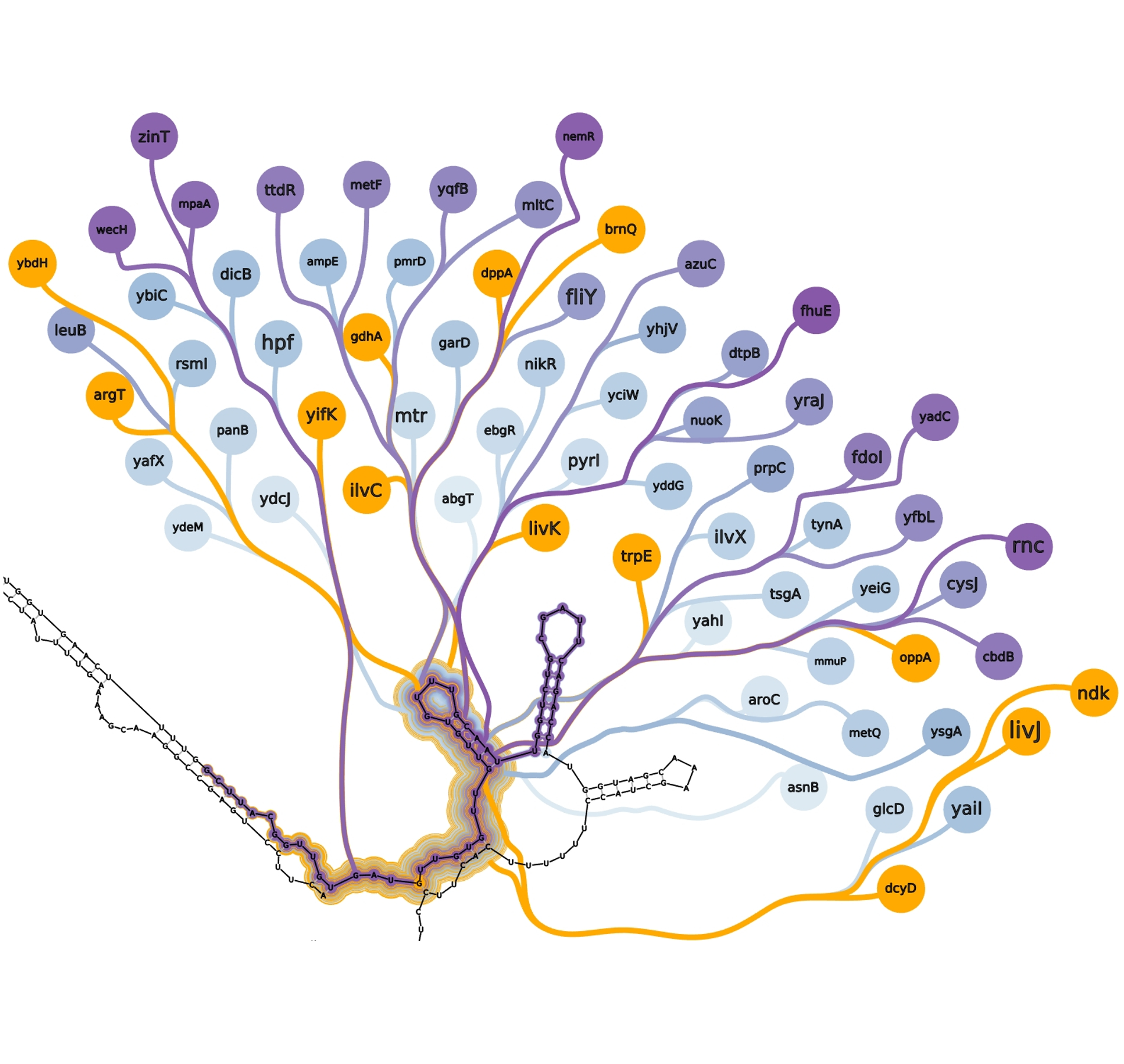
Ces travaux ont constitué le point de départ pour la représentation de données relationnelles massives et, par application des résultats obtenus, ont amené à la création d'une nouvelle technique de visualisation associée à des outils d'interaction et d'exploration multi-échelle.
Cette méthode de visualisation a ensuite été testée sur un cas d'application avec des données représentant des visites d'utilisateurs sur un site web dans le cadre d'une architecture Big Data (stockage distant, calculs parallélisés et distribués, visualisation déportée).
L'objectif ici était de fournir une représentation interactive à différents niveaux de détails des visites d'utilisateurs, et plus exactement d'identifier les provenances et destinations dans le parcours des utilisateurs passant par une catégorie de pages particulière.
Ce travail a été gratifié du Best Paper Award dans sa catégorie lors de sa présentation à la conférence internationale Information Visualisation en 2015 [2].
Le deuxième aspect de ces recherches a été dédié à la visualisation de données multi-dimensionnelles.
Une façon de se représenter ce type de données consiste à imaginer un tableau dont les lignes sont les entités et les colonnes les attributs des entités, chaque case du tableau correspond ainsi à la valeur d'une entité donnée pour une propriété donnée.
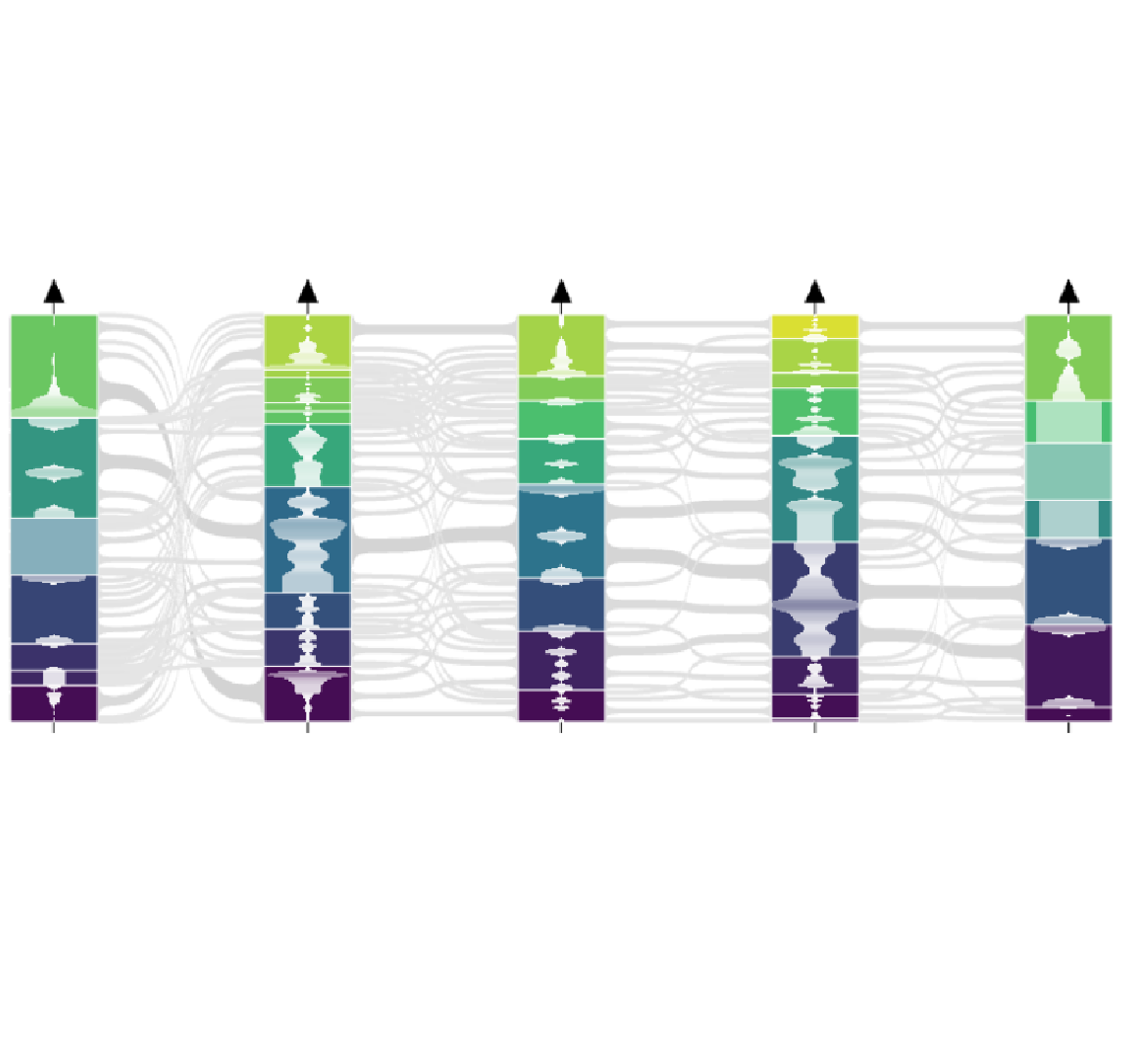
Une technique de visualisation couramment utilisée pour ce type de données est celle des coordonnées parallèles et consiste à attribuer un axe vertical par attribut (l'axe étant gradué pour un attribut numérique) et à tracer, pour chaque entité du tableau, une ligne qui croise chaque axe à la valeur correspondante pour l'entité considérée.
Cette technique, bien que particulièrement adaptée pour la recherche de corrélations, anti-corrélations ou motifs divers dans les données, est particulièrement sensible à la quantité d'entités à représenter\,: plus elle est importante moins la technique est efficace, chaque ligne tracée ayant un impact direct sur le temps nécessaire pour créer ou interagir avec la représentation mais aussi sur sa lisibilité.
Les réflexions pour répondre à ces limitations et par généralisation des travaux précédents ont conduit à la mise en place d'un système d'analyse visuelle de données massives prenant en compte le stockage, le calcul et le transfert des données.
Ainsi il devient possible, malgré une architecture Big Data, de visualiser et explorer de façon interactive des données multi-dimensionnelles pouvant atteindre plusieurs milliards d'éléments en temps réel avec des coordonnées parallèles abstraites déportées [5].
Un troisième aspect, conduit en parallèle aux deux premiers, consiste à créer et tester les abstractions visuelles pour des données biologiques.
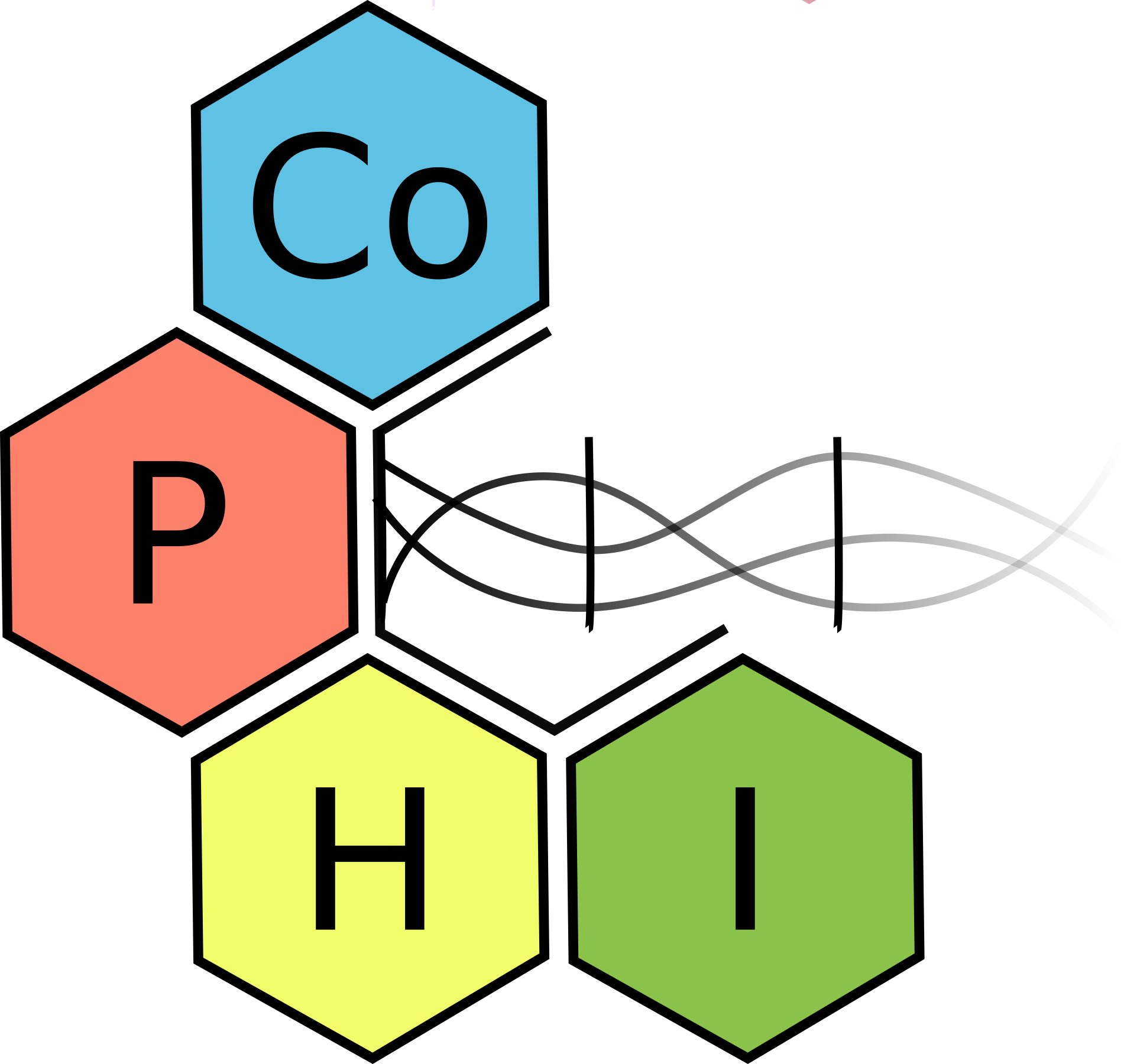
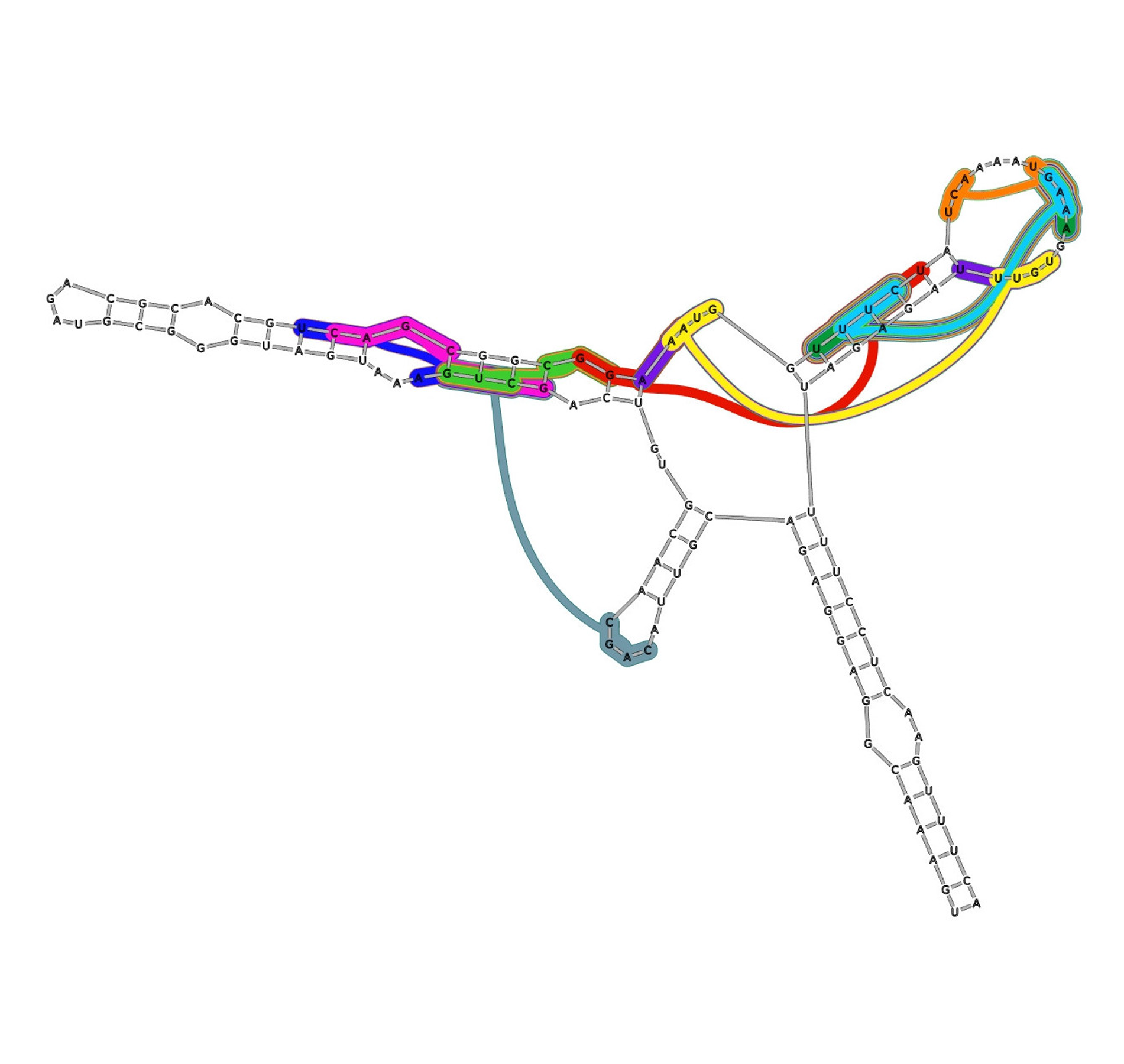
Une première partie sur la visualisation de données relationnelles issues d'analyse en génomique a permis de représenter les probabilités d'appariements des bases nucléiques dans la structure secondaire d'ARN [4] ainsi que des prédictions d'appariements entre petits-ARN et ARN-messagers [3].
Une seconde partie consiste en un cas d'étude et d'application des coordonnées parallèles abstraites pour la recherche de corrélations dans des analyses en génomique et protéomique.