Quel plaisir d’assister (en tant qu’invité), à la thèse de Maxime Mahout, intitulée “Programmation logique pour l’analyse des flux métaboliques et applications à la biologie”, encadrée par Sabine Pérès. C’est une thèse dans laquelle je suis investi depuis le stage de Master 1 (et oui !) de Maxime, au travers de 5 ans de discussions, de visios sous covid, d’idées loufoques, de moments de doutes et de découvertes (pour ma part) de mécanismes cellulaires et de maladies effrayantes. L’idée est de contrebalancer l’incroyable complexité combinatoire des mécanismes de la cellule (et même de consortium de cellules) par la force du raisonnement logique et par contraintes. Grâce à une spécialisation d’un moteur ASP, maxime a pu proposer des pistes de traitement pour des maladies encore dramatiques en faisant preuve d’une persévérance à toute épreuve.
Nous avons donc pu profiter du formidable travail de modélisation des biologistes, rendant maintenant accessible ces mécanismes à l’oeuvre dans les cellules par la logique (ou sous forme de graphes). Cela permet ainsi de pouvoir raisonner sur les mécanismes à l’oeuvre dans les cellules, vues comme des usines à métabolites, produisant ainsi des protéines à partir de règles chimiques clairement identifiées. Une fois cette modélisation faite, de nombreux problèmes peuvent être formulés en logique (quel gène desactiver pour empêcher la cellule de produire tel enzyme, tout en conservant ses bonnes propriétés ?), ouvrant ainsi la voie à de nouvelles possibilités d’analyses.
Je ne peux que vous conseiller de lire la thèse, ou de vous référer à l’article que nous avons pu publier dans Nature Processing Journal (Systems Biology and Applications), en suivant ce lien.

Les travaux de thèse de Maxime permettent donc, par exemple, d’envisager des thérapies pour des infections extrêmement graves, causant des milliers de décès et d’amputations. Il a ainsi identifié ce qui est particulier lorsque deux bactéries (que l’on peut traiter individuellement) collaborent pour survivre. Lorsque que P. aeruginosa et S. aureus colonisent une plaie, il est quasi impossible de traiter l’infection, le consortium devient extrêmement résistant à tous les traitements antibiotiques. Dans ses travaux, Maxime parvient même à identifier des voies métaboliques à inhiber pour potentiellement bloquer le consortium en entier.
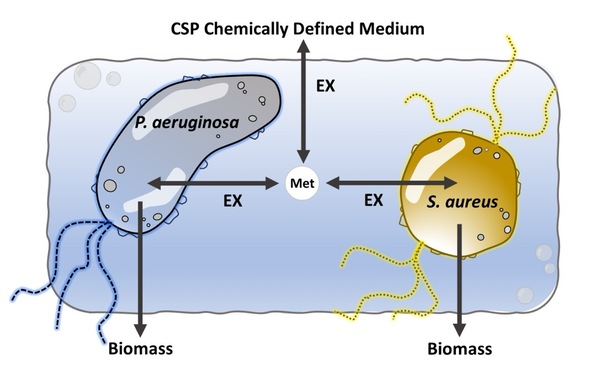 Une illustration des bactéries P. aeruginosa et S. aureus, qui, lorsqu’elle collaborent, deviennent absolument redoutables à combattre
Une illustration des bactéries P. aeruginosa et S. aureus, qui, lorsqu’elle collaborent, deviennent absolument redoutables à combattre
Cela a été pour moi l’occasion de toucher du doigt l’importance et la difficulté de la modélisation en biologie, l’extrême combinatoire des réseaux métaboliques et la force du raisonnement logique, seul capable d’appréhender cette complexité pour y voir clair, dans un cadre loin de la résolution d’instances aléatoires ou jouets, qui sont souvent les principaux objets en face du chercheur très en amont.
Bravo encore Maxime !